鳓鱼(Ilisha elongata)隶属于鲱形目(Clupeiformes),锯腹鳓科(Pristigasteridae),鳓属(Ilisha),是一种广泛分布于印度洋和太平洋西部的暖水性近海中上层的经济性洄游鱼类,在中国的渤海、黄海、东海、南海均有分布。因其重要的经济价值和广泛的地理分布,鳓鱼成为了研究生物适应性演化的理想模型。随着全球气候变化、栖息地破坏和强捕捞作业的压力,鳓鱼已呈现个体小型化和性成熟提早的趋势,资源出现严重衰退。开展鳓鱼全基因组的研究对有效管理和保护鳓鱼种质资源尤为重要。本研究以鳓鱼为研究对象,对其进行全基因组测序和组装,通过全基因组的构建和比较基因组学分析,深入解析鳓鱼全基因组结构和染色体水平基因组进化演化特征。研究结果近日以“Chromosome-level genome assembly and annotation of Chinese herring (Ilisha elongata)”为题目发表在《Scientific Data》期刊上。
研究的主要结果包括:
1、使用二代MGI和三代PacBio两种高通量测序平台,对鳓鱼的全基因组进行了高精度的测序工作。通过Hi-C技术辅助组装将contig序列锚定到24条染色体上,构建了一个连续性高、覆盖率好的高质量染色体级别的鳓鱼基因组图谱。最终,完成的鳓鱼基因组大小约为815 Mb,contig N50为4.82 Mb,scaffold N50为32.61 Mb,染色体挂载率为95.32%;SNP和InDel纯合率分别为0.003%和0.012%,经过BUSCO评估,基因组组装的完整性高达96.68%,这表明基因组组装的质量较高,这为后续的遗传演化和比较基因组分析奠定了坚实的基础。
2、对组装完成的鳓鱼基因组进行了全面的注释,通过从头预测和同源预测的方式进行重复序列注释,得到的重复序列为295.7 Mb,占整个鳓鱼基因组的35.08%。通过转录组预测得到25,923个基因,转录组比对率高达99.05%,表明转录组数据与基因组序列的匹配度非常高;利用同源蛋白的信息,使用GeneWise预测到33,951个基因;使用AUGUSTUS基于隐马尔可夫模型预测到46,804个基因。最终,基于从头预测,同源预测和RNAseq预测,总共预测得到26,381个蛋白质编码基因,其中24,596个基因得到了功能注释,这意味着这些基因可能参与了已知的生物学过程或具有可识别的蛋白质功能域。而那些未能成功注释的基因,可能代表了鳓鱼特有的基因,这些基因的功能和特性有待进一步的研究探索。
综上所述,本研究首次对鳓鱼进行了全基因组测序和组装,并成功组装出了一个染色体级别的高质量基因组参考图谱,为后续研究提供了坚实的基础。此外,本研究对鳓鱼基因组进行了全面的注释工作,并通过比较基因组学分析深入探讨了鳓鱼的演化历程。这些成果为鳓鱼及其近缘鲱形目鱼类的生物学和遗传学领域研究提供了丰富的基因组数据。总的来说,该研究的结果为鳓鱼资源的科学管理与可持续发展提供了理论依据,同时也为探究鳓鱼基因组特征和遗传学解析打下了坚实的研究基础。
原文链接:https://www.nature.com/articles/s41597-025-04790-7
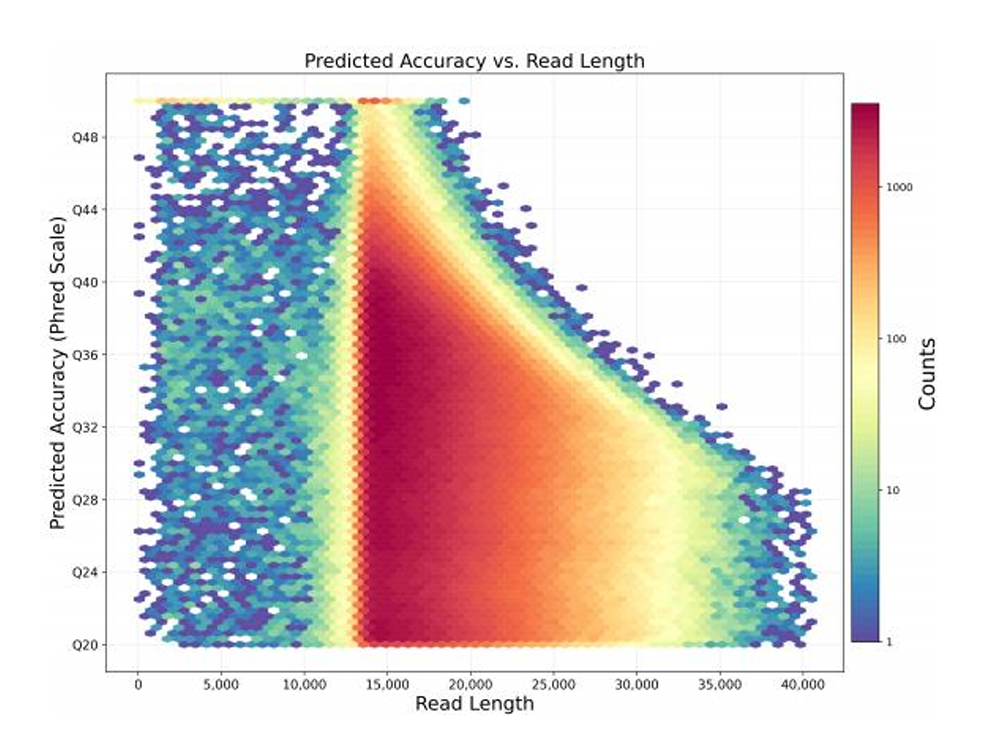
图1鳓鱼基因组三代 PacBio 测序结果质量与长度 2D 统计图
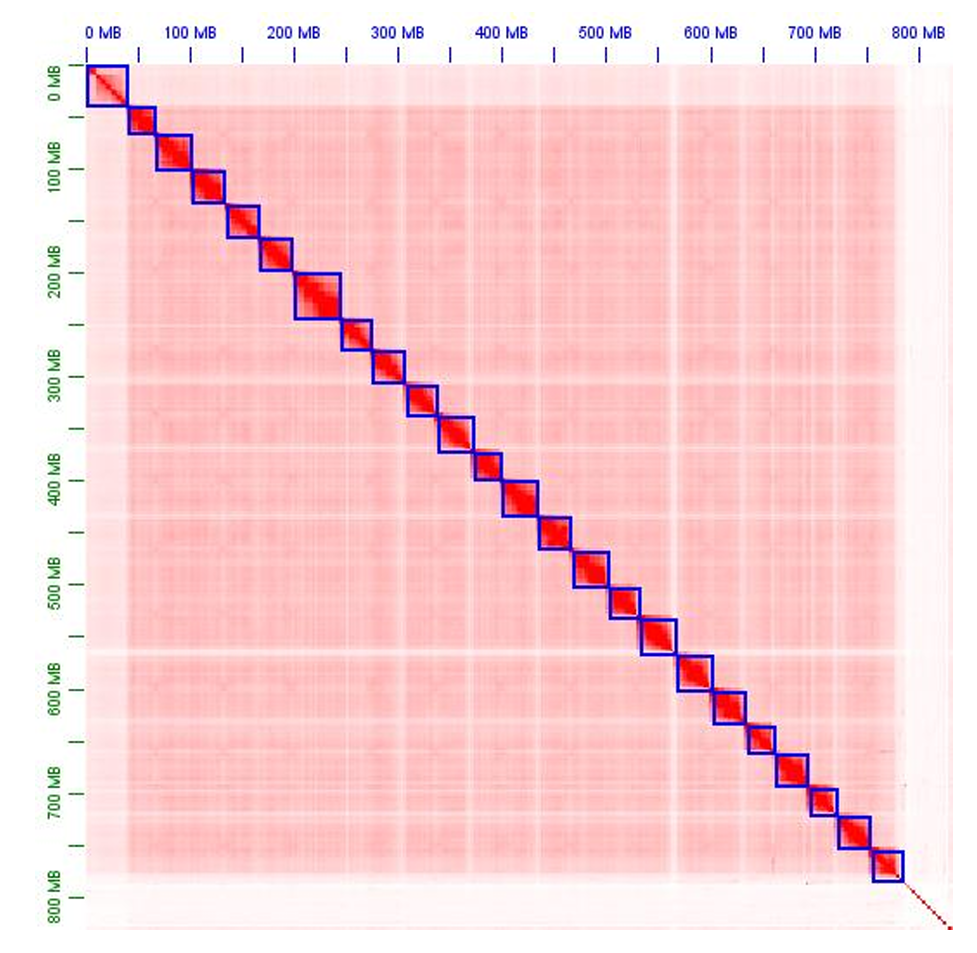
图2鳓鱼染色体基因组的HI-C相互作用热图
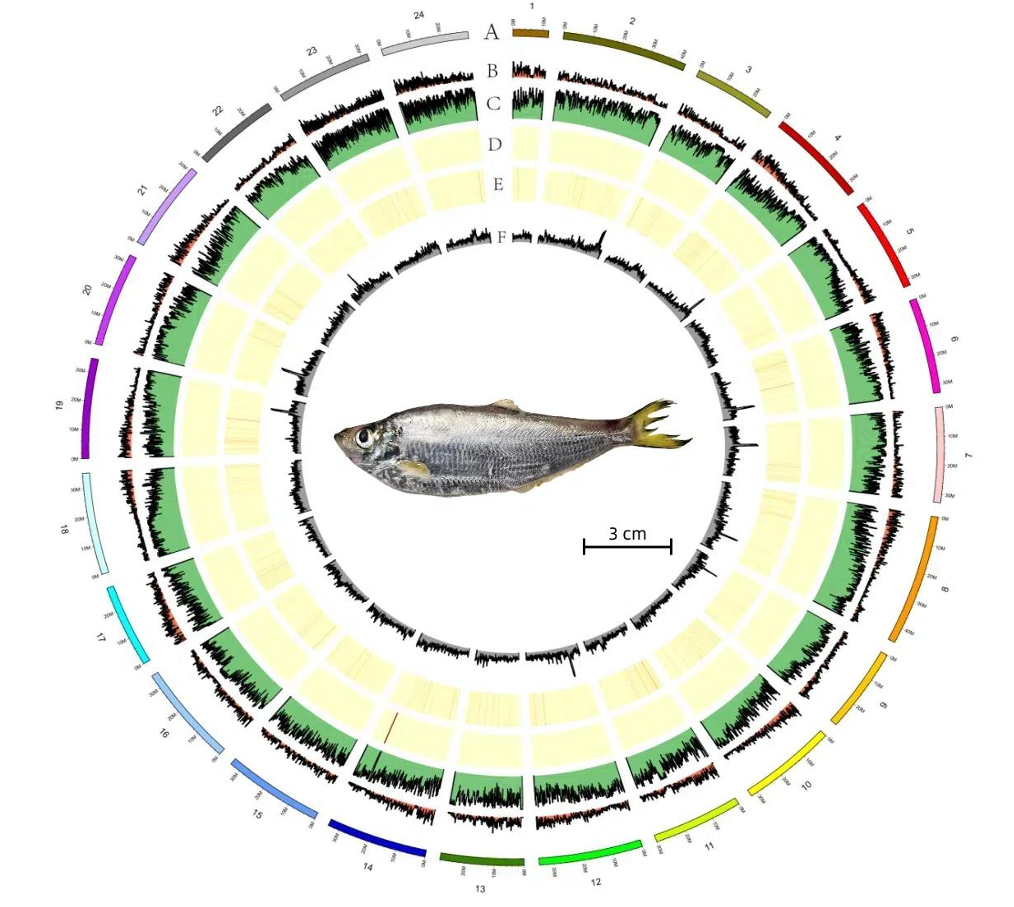
图3鳓鱼染色体水平基因组圈图
注:(A)染色体长度,(B)GC 含量分布,(C)二代测序深度分布,(D) 三代测序深度分布,(E)基因密度分布,(F)长串联重复序列分布。
